Les deux méthodes de diagnostic les plus couramment utilisées pour détecter le SDRP sont l'ELISA et la PCR. Dans cet article, nous allons nous concentrer sur la PCR, la méthode par laquelle le virus peut être détecté dans pratiquement n'importe quel matériel. Il est basé sur la polymérisation de l'ADN cyclique d'un ADN ou, comme dans le cas du virus SDRP, de fragments d'ADNc flanqués de courtes séquences auxquelles des amorces d'ADN spécifiques sont attachées à chaque cycle de PCR. La sensibilité et la spécificité d'un test de diagnostic dans lequel la PCR est utilisée dépendent de l'adéquation de ces amorces à leur objectif et des cycles thermiques utilisés pour l'amplification cyclique. Idéalement, elles ne devraient être complémentaires que du génome d'une espèce virale spécifique et ne pas pouvoir se lier à un autre microbe ni à une séquence d'ADN hôte, tout en étant capables de détecter les génomes de toutes les souches ou de tous les variants de cette espèce de virus. Par conséquent, les fragments les plus conservés du génome sont leur objectif.
Le virus du SDRP est l’un des agents pathogènes pour lesquels il est plus difficile de mettre au point des tests PCR très sensibles et spécifiques. Les virus SDRP actuellement connus appartiennent à deux espèces, SDRPv-1 et SDRPv-2. Les deux espèces de virus du SDRP se sont répandues dans le monde entier, mais le SDRPv-1 prédomine en Europe, alors que le SDRPv-2 est prédominant en Amérique et en Asie. On peut supposer que la diversité génétique de l'espèce de virus prédominante est supérieure à celle de l'espèce dont la prévalence est la plus faible. En effet, les tests PCR utilisés, par exemple, en Europe ou en Amérique, peuvent être mieux validés et mieux adaptés à la détection du SDRPv-1 ou du SDRPv-2, respectivement. En conséquence, certaines souches de l'espèce "secondaire" dans une région ou un continent donnés peuvent ne pas être détectées, ce qui sous-estimerait leur rôle dans la pathogénie du SDRP. Ceci est particulièrement important dans les élevages où cohabitent SDRPv-1 et SDRPv-2. La grande diversité génétique des virus SDRP nécessite une mise à jour constante des séquences des amorces et des sondes PCR afin de suivre l'évolution continue du virus et la découverte de variants génétiques complètement nouveaux. Ceci est particulièrement vrai pour le SDRPv-1, dont l'extrême diversité génétique, associée à la faible disponibilité de virus de référence en provenance de Russie, de Biélorussie et d'Ukraine, menace d'affecter la sensibilité des kits PCR commerciaux pour les sous-types génétiques de SDRPv-1. en Europe de l'Est.

Actuellement, la variante PCR en temps réel est principalement utilisée dans les diagnostics de routine. Il existe plusieurs méthodes PCR en temps réel dont les principes diffèrent, mais la plus utilisée est la méthode avec les sondes TaqMan. Une sonde TaqMan est un court fragment d'ADN (oligonucléotide) marqué avec des fluorophores, qui se lie à l'ADN cible entre les amorces adjacentes. Si une liaison se produit, la sonde est lissée pendant la polymérisation de l'ADN et l'appareil de PCR enregistre la fluorescence. La PCR en temps réel est particulièrement adaptée au multiplexage des objectifs de diagnostic. Cela signifie que dans une seule réaction PCR (ou dans un seul test), 2 à 3 agents pathogènes différents peuvent être détectés par l'émission de fluorescence de 2 à 3 couleurs différentse. Par conséquent, les deux espèces de virus du SDRP peuvent être facilement détectées en même temps et différenciées dans des échantillons d'un seul animal, case ou élevage.
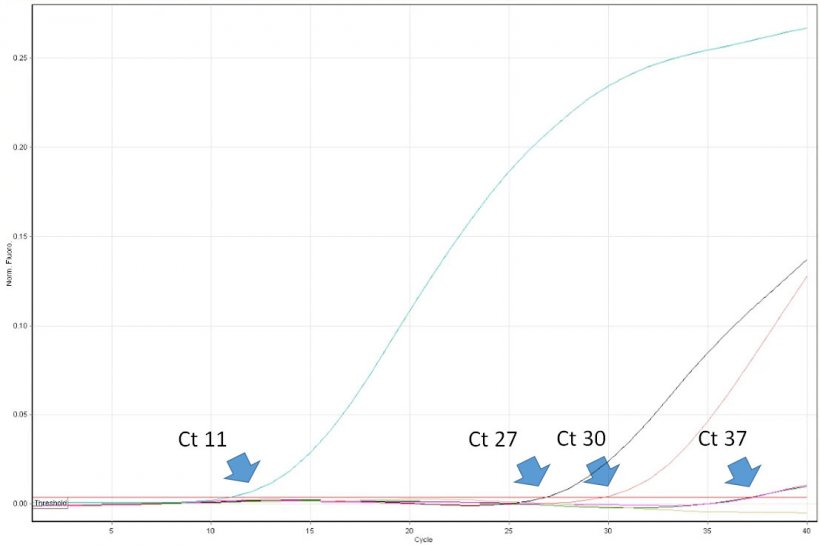
Figure 1. Le cycle seuil (Ct) est le premier numéro du cycle d'une PCR en temps réel dans lequel la fluorescence qui indique la présence de l'agent pathogène souhaité dans l'échantillon est détectée. Plus la valeur de Ct est basse, plus la quantité d'agent pathogène dans l'échantillon analysé est grande. Les valeurs très élevées de Ct doivent être interprétées avec prudence car elles peuvent provenir de la dégradation spontanée d'une sonde TaqMan au cours des derniers cycles, malgré l'absence d'ADN cible dans l'échantillon.
L'interprétation des résultats de la PCR est d'une simplicité trompeuse. Le vétérinaire doit connaître les limites de la méthode et pouvoir interpréter les résultats de manière critique. Par exemple, un résultat PCR positif démontre la présence d'un acide nucléique cible (par exemple, du virus SDRP) dans l'échantillon. Cependant, cela ne prouve pas que l'échantillon contient des virus infectieux. La PCR en temps réel permet la quantification du virus dans l'échantillon. Leurs résultats sont exprimés en "cycle seuil" (Ct), qui est le premier numéro de cycle dans lequel l'appareil de PCR a détecté l'émission de fluorescence par le tube de réaction. Plus le nombre de cycles est petit, plus le nombre de copies de l'ADN cible est élevé (lu comme le nombre de copies de virus). Par exemple, un échantillon avec un Ct 20 peut être considéré comme hautement positif, et un échantillon avec un Ct 35 faiblement positif. Cependant, dans certains cas, la fluorescence n'est détectée que lors de cycles très tardifs de PCR en temps réel, par exemple,> 38. Comment peut-on interpréter de telles valeurs ? Malheureusement, ce résultat n'indique pas toujours la présence d'un très petit nombre de copies de virus dans l'échantillon. Les "virus à croissance tardive" ainsi dénommés peuvent être le résultat de la rupture spontanée d'une sonde après de nombreux cycles thermiques de PCR, qui simulent la nucléolyse enzymatique provoquée par l'ADN polymérase. Par conséquent, cela peut également se produire en l'absence de l'acide nucléique du virus dans l'échantillon. L'importance d'une interprétation correcte de la présence de «virus à croissance tardive» est maximale lors du suivi de populations sans virus SDRP. Un diagnostic de suivi doit être établi, incluant la technique ELISA. Malheureusement, certains laboratoires de diagnostic en Europe ne fournissent pas aux clients les valeurs de Ct précises de la PCR en temps réel. Au contraire, les résultats ne sont exprimés que comme négatifs ou positifs. L'utilité de tels rapports pour les professionnels est limitée et, dans certains cas, peut conduire à des décisions erronées concernant les protocoles de contrôle du virus du SDRP.
Considérations pratiques
- Aucune des méthodes de PCR actuelles ne peut détecter tous les virus du SDRP.
- Le résultat négatif d'une PCR provenant de quelques échantillons aléatoires d'un élevage ne prouve pas la négativité pour l'infection SDRP.
- Le résultat positif d'une PCR démontre la présence d'acide nucléique SDRPv dans un échantillon, mais pas nécessairement son pouvoir infectieux.









