La détermination du sérotype d'Actinobacillus pleuropneumoniae (App) est importante pour son contrôle, car différents sérotypes ont un potentiel de virulence différent (selon la zone géographique), et cette information peut être utilisée pour choisir le vaccin le plus approprié (Gottschalk, 2015) . Sur la base de sa capsule, il existe 18 sérotypes d'App connus (Bossé et al., 2018a), 1, 5, 9 et 11 étant les plus virulents. Sur la base des besoins de NAD (nicotinamide adénine dinucléotide), les isolats du biotype 1 sont différenciés de ceux du biotype 2, ou NAD-indépendants.
Classiquement, des tests sérologiques comprenant l'agglutination, la coagglutination, l'immunodiffusion, l'hémagglutination indirecte et la précipitation en anneaux ont été utilisés pour déterminer le sérotype. Cependant, des antisérums spécifiques avec des titres élevés sont nécessaires, et les réactions croisées entre les sérotypes (par exemple 3/6/8 et 1/9/11) sont un problème. Nous avons constaté que la majorité des isolats du Royaume-Uni et d'Irlande assignés sérologiquement au sérotype 3 appartenaient au sérotype 8 (O'Neill et al., 2010). En raison de ces problèmes, nous avons développé des tests moléculaires utilisant la réaction en chaîne de la polymérase (PCR) pour amplifier des séquences d'ADN spécifiques de chaque sérotype trouvé dans les gènes de biosynthèse de la capsule (Bossé et al., 2014). L'utilisation de ces séquences spécifiques de chaque sérotype a permis de développer un test spécifique pour le sérotype 16 (Bossé et al., 2017), et a conduit à la découverte récente des sérotypes 17 et 18 (Bossé et al., 2018a). Avec la découverte des sérotypes 16-18, nous avons décidé de produire un test PCR qui pourrait déterminer tous les sérotypes connus d'App. Cependant, les sérotypes 9 et 11 ne peuvent pas être différenciés par PCR, car leurs loci capsulaires sont pratiquement identiques (Bossé et al., 2018b), et ils ne peuvent pas non plus être déterminés par des tests sérologiques (Gottschalk, 2015). Le test PCR devait confirmer l'isolat en tant qu'App et déterminer le sérotype spécifique. Pour confirmer App, nous avons utilisé des amorces qui ont amplifié une région de 418 pb du gène apxIV, spécifique à App (Schaller et al., 1999). Cependant, en raison du grand nombre de sérotypes (n = 18), nous avons dû formuler deux PCR multiplexes (mPCR), chacune capable de détecter de nombreux sérotypes. MPCR1 détecte les sérotypes 1-12 et 15 (figure 1A) et mPCR2 les sérotypes 13-14 et 16-18 (figure 1B). Les isolats qui amplifient seulement une bande apxIV dans le mPCR1 sont ensuite analysés avec le mPCR2.

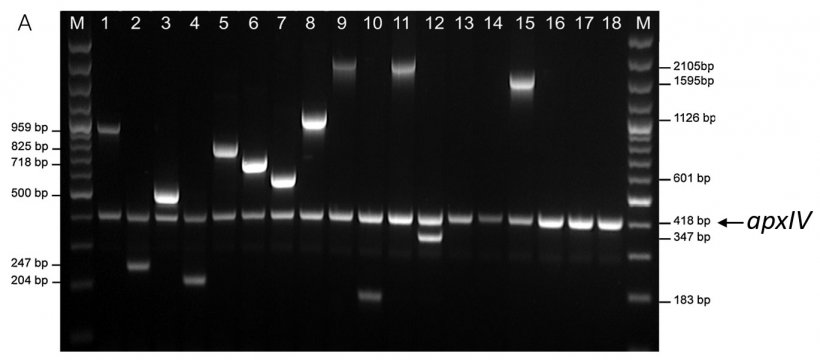
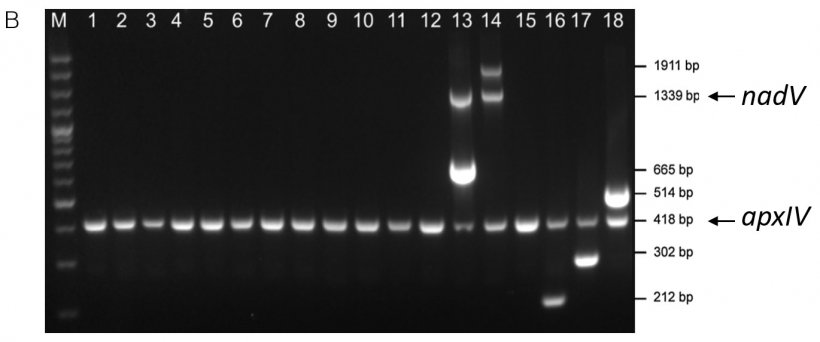
Figure 1. Détection spécifique des sérotypes d'amplicons des souches de référence d'App par (A) mPCR1 pour les sérotypes 1-12 et 15 et (B) mPCR2 pour les sérotypes 13-14 et 16-18. Sérotype des souches de référence 1-18 = lignes 1-18. Les deux mPCR montrent un amplicon apxIV de 418 pb qui confirme que les échantillons sont App (adapté de Bossé et al., 2018b).
mPCR2 confirme également le biotype par des amorces conçues pour amplifier un fragment de 1339 pb du gène nadV (qui confère l'indépendance au NAD). L'amplicon nadV de 1339 pb n'est détecté que dans les souches de référence du biotype 2 (sérotypes 13-14). Il convient de noter que d'autres sérotypes (par exemple 2, 4, 7 et 17) ont été décrits comme appartenant au biotype 2, et certains sérotypes d'isolats nord-américains du sérotype 13 comme appartenant au biotype 1 (Gottschalk, 2015).
La matrice d'ADN pour les PCR peut être de l'ADN purifié (soit à partir de bactéries cultivées ou d'échantillons de tissus) obtenu à l'aide de kits commerciaux, de lysats de cellules bactériennes entières bouillies ou de colonies d'une culture sur plaque. L'ADN purifié fournit de meilleurs résultats, tandis que les PCR de colonies peuvent donner des résultats négatifs partiels / faux négatifs si les colonies sont très collantes et difficiles à lyser, comme le montrent les trois isolats cliniques de sérotype 2 qui ont seulement amplifié la bande spécifique de sérotype de PCR de colonie, mais qui ont amplifié à la fois celle-ci et la bande spécifique d'apxIV en utilisant de l'ADN purifié (figure 2). En de rares occasions, les isolats ne pourront pas amplifier l'apxIV, même en utilisant de l'ADN purifié (Bossé et al., 2014). Lorsque cela se produit, d'autres amorces d'apxIV (oAPXIV-TSP1 / 2) peuvent être utilisées pour confirmer l'App (Tegetmeyer et al., 2008). mPCR1 et mPCR2 peuvent être utilisés pour identifier de nouveaux sérotypes, comme nous l'avons fait avec les 17 et 18 (Bossé et al., 2018a). Les isolats d'App qui produisent une bande apxIV mais pas des amplicons spécifiques de sérotype peuvent potentiellement être un nouveau sérotype, bien que l'absence d'une bande spécifique de sérotype puisse être due à un manque de convergence de l'amorce sur des isolats divergents ou à la présence d'un élément d'insertion qui modifie le locus de la capsule. Le séquençage du génome complet de l'isolat confirmera laquelle de ces options est correcte.
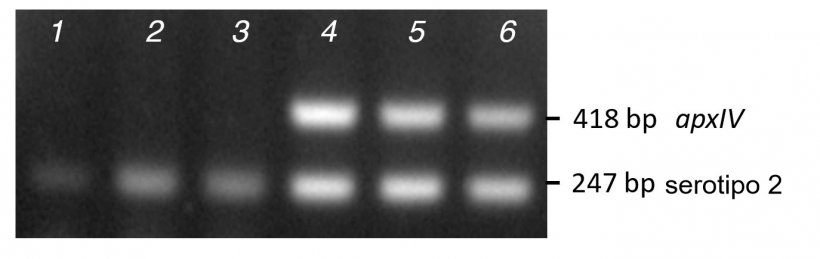
Figure 2. Comparaison de l'amplification de bandes à partir de PCR de colonies purifiées (lignes 1-3) par rapport à l'ADN (lignes 4-6) pour trois isolats cliniques de sérotype 2 en utilisant le mPCR1.
En résumé, la surveillance des sérotypes d'APP dans un élevage ou un pays est importante pour contrôler la maladie. Nos PCR de sérotypage (mPCR1 et mPCR2) sont des outils utiles pour identifier les sérotypes virulents, les bons vaccins à utiliser (commerciaux ou autovaccins), et pour empêcher l'introduction de porcs avec des souches d'App potentiellement virulentes dans les élevages non exposés. De plus, notre méthode a le potentiel pour identifier de nouveaux sérotypes d'App et améliorer les diagnostics.
Remerciements
La recherche sur APP dans le laboratoire des auteurs est soutenue par le Biotechnology and Biological Sciences Research Council du Royaume-Uni.




