Les infections par le virus de l'influenza A (IA) présentent une morbidité significative dans les populations porcines du monde entier. L'infection est associée essentiellement à des symptômes respiratoires aigus qui souvent sont accompagnés de coïnfections bactériennes qui peuvent donner lieu à une augmentation de la mortalité chez les porcelets, à une réduction de la croissance en engraissement ou à des avortements chez les truies gestantes. Pour toutes ces raisons, les pertes économiques peuvent être considérables (Rajao et al., 2014 ; Vincent et al., 2014 ; Vincent et al., 2014).
Les virus influenza A porcins font partie de la famille Orthomyxoviridae ; ce sont des virus à ARN monocaténaire dont le génome compte 8 segments. Les sous-types sont classés d'après les caractéristiques génétiques et antigéniques des glycoprotéines de surface hémagglutinine (HA) et neuraminidase (NA) (Cheung et al., 2007).

Les fractions glycanniques avec des acides sialiques terminaux agissent comme des récepteurs pour l'attachement du virus IA aux cellules superficielles des voies respiratoires. À cause de l'expression des récepteurs avec des acides sialiques terminaux liés en α-2-6 et α-2-3 dans les voies respiratoires supérieures des porcs, ceux-ci sont susceptibles au virus IA d'origine aviaire et humaine aussi (Byrd-Leotis et al., 2014 ; Trebbien et al., 2011 ; Walther et al., 2013). Par conséquent, les coïnfections de cellules porcines avec des virus IA aviaires et/ou humains ouvrent la porte à d'éventuels réarrangements génétiques (échanges de segments de génomes) entre ces virus (Hass et al., 2011 ; Ma et al., 2009 ; Van Poucke et al., 2010 ; Zell et al., 2013). Ces réarrangements peuvent supposer des changements phénotypiques inattendus ou difficiles à prédire influant sur le tropisme des hôtes et des tissus, la virulence et la composition antigénique, par rapport à leurs virus parentaux.
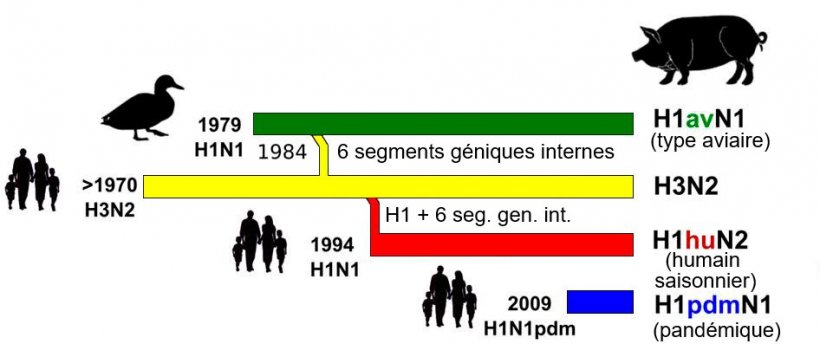
Figure 1. Histoire et origine des virus d'influenza porcine type A (IA) qui circulent actuellement en Europe. Veuillez noter qu'aucun de ces virus IA n'a leur origine dans le porc.
La figure 1 montre l'origine et l'histoire du réarrangement des virus IA porcins qui circulent actuellement dans les populations porcines européennes. Jusqu'en 2009, trois sous-types de virus IA, H1N1, H1N2 et H3N2, circulaient en Europe. La lignée la plus ancienne, le H1N1, s'est introduite dans la population porcine à la fin des années 1970 à partir d'une origine aviaire et a remplacé la lignée H1N1 type classique dérivée de la lignée humaine de 1918 qui était en circulation au cours des années précédentes. S'agissant d'un virus influenza d'origine aviaire, la lignée H1N1 après les années 70 est appelée H1N1 "type aviaire" (ou "av") (Brown, 2013 ; Simon et al., 2014). En 1984, un réarrangement a eu lieu entre un virus saisonnier humain H3N2 et le sous-type H1avN1av dont le virus qui en résulte contient tous les 6 segments internes d'origine aviaire et les HA (H3) et NA (N2) du virus humain (Kong et al., 2015). Ce nouveau virus s'est établi comme endémique dans les populations porcines de plusieurs pays européens mais apparemment pas dans tous les pays. En 1994, un nouvel réarrangement a affecté le virus H3N2 porcin, qui a échangé son segment de gène HA avec celui d'un autre virus saisonnier humain H1N1 pour générer la lignée H1N2. Ce sous-type H1 est appelé H1N2 "type humain" (ou "hu") (Brown, 1998). Ces lignées ont circulé d'une manière relativement stable dans les pays européens avec certaines restrictions géographiques par rapport à leur prévalence (Brown, 2013 ; Kuntz-Simon et al., 2009). Les virus de la lignée H1N1av semblaient être les responsables de la plupart d'infections chez les porcs en Europe (Brown, 2000).
En 2009, un quatrième acteur entre en jeu : le virus influenza A H1N1 pandémique de 2009 (H1N1pdm). Ce virus est le résultat de multiples réarrangements et comporte des segments originaires de virus américains de type aviaire (PB2, PA), humain (PB1) et porcin classique (HA, NP, NS), et de virus européens de type porcin (NA, NP) (Neumann et al., 2009). Par rapport à l'origine au moins partiellement porcine de cette dernière pandémie de grippe, l'infection chez les humains a été appelée "grippe porcine". Les porcs semblaient être très susceptibles à ce type de virus IA et la transmission zoonotique inverse de l'homme au porc a été enregistrée dans plusieurs endroits du monde entier. Depuis, des infections par H1N1pdm ont lieu dans des populations porcines dans des cycles qui sont indépendants des infections chez l'homme (Brookes et al., 2010).
Suite à ce qui a été appelé “grippe porcine”, un programme de surveillance de l'influenza porcine a été mis en place en Europe de 2013 à 2015, à travers le projet ESNIP3 (Watson et al., 2015). Les résultats ont montré que le H1N1dpm est présent dans plusieurs pays européens et, en particulier, avec une prévalence élevée en Grande-Bretagne et en Irlande. La présence d'H1N1dpm d'origine humaine dans les populations porcines européennes a bien évidemment altéré l'équilibre minutieux entre les trois lignées authentiques de virus d'influenza porcins européens et a entraîné l'augmentation d'événements de réarrangement. Parmi les virus émergents dérivés des réarrangements des segments H et/ou N, la lignée H1pdmN2 est apparue en Allemagne, laquelle circule, à faible prévalence, depuis des années maintenant (Starick et al., 2012 ; Watson et al., 2015). La protéine HA de ce virus émergent a subi des changements significatifs, et elle est antigéniquement distinguable de l'H1pdm d'origine humaine (Harder et al., 2013). D'autres réarrangements ont été décrits (H1huN1av, H1dpmN1av, H3N1pdm) qui n'ont pas été capables de maintenir leur circulation (Watson et al., 2015). En plus, d'autres réarrangements ont également été décrits, lesquels ont échangé tous ou certains des six segments internes du génome avec des virus H1N1pdm (Kong et al., 2015 ; Watson et al., 2015). Aux É.-U., les virus H3N2 ou H1N1 porteurs du segment M de l'H1N1pdm ont été associés à une plus grande transmission du porc à l'homme dans des foires agricoles (Schicker et al. 2016). Ces “variantes” de virus ont également été sporadiquement signalées en Europe mais elles n'ont pas été associées à des infections chez l'être humain. La prévalence de l'H1N1dpm dans les populations porcines européennes varie considérablement (figure 2). Contrairement à la situation dans les îles britanniques, le sous-type porcin H1N1av présente toujours la prévalence la plus élevée de l'Europe continentale, alors que les sous-types H3N2 et H1huN2 présentent des prévalences inférieures (figura 2). Le sous-type H3N2 n'a pas été détecté au Danemark, en Pologne ou le Royaume-Uni, et seuls quelques foyers sporadiques ont été déclarés en France (Watson et al. 2015).
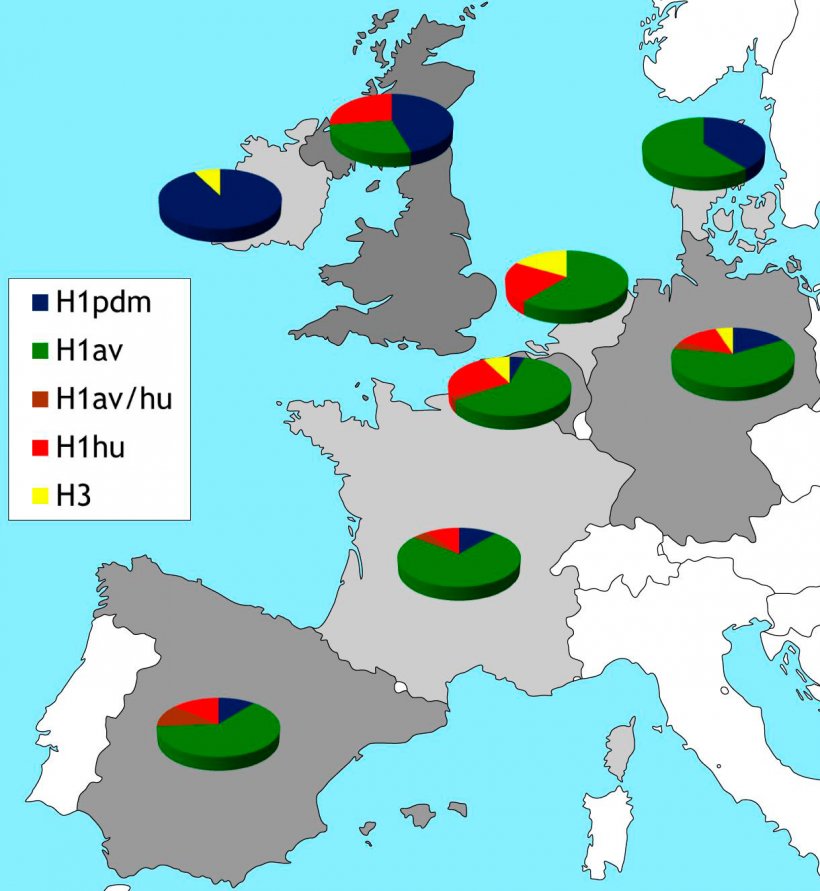
Figure 2. Restrictions géographiques des sous-types HA des virus IA qui circulent actuellement en Europe. Les données sont basées sur (références) et sur nos études de surveillance passive lancées en 2015.
Des études de surveillance passive ont été lancées dans plusieurs pays de l'Europe centrale et de l'est après la fin du projet ESNIP3 en 2015, lesquelles sont établies dans notre laboratoire. Les données préliminaires de la première année de surveillance montrent une fréquence plus élevée de l'H1N1pdm et de ses réarrangements. En plus, on constate une plus grande fréquence de coïnfections et une plus grande activité de réarrangement entre les trois lignées H1 porcines européennes qui sont en circulation. Reste à clarifier dans quelle mesure ces conclusions sont biaisées par la mise en place d'outils de diagnostic précis développés par notre laboratoire : la détection du virus IA porcin et la caractérisation de lignées et de sous-types HA (H1av, H1hu, H1pdm, H3) et NA (N1av, N1pdm, N2) sont réalisées à travers un ensemble de trois RT-qPCR à temps réel. La plus grande sensibilité de ces tests permet une analyse directe d'échantillons de terrain (en particulier, des prélèvements nasaux) indépendamment de l'isolement des virus dans les cultures cellulaires (Henritzi et al., 2016). Ces études se poursuivront au moins jusqu'à la fin de 2017.
La surveillance continue des populations porcines en Europe est essentielle pour le suivi de l'évolution du virus IA porcin. Ce virus peut jouer un rôle important dans la génération de virus pandémiques chez l'être humain, comme l'apparition du virus H1N1 de 2009 l'a prouvé. La meilleure connaissance des changements génotypiques dynamiques et l'apparition de nouveaux phénotypes du virus IA ayant une propension zoonotique potentiellement augmentée sont au centre de la politique OneHealth.
Remerciements
Les études récentes sur le virus IA porcin dans le laboratoire des auteurs ont été financées par IDT, Dessau, Allemagne. Les auteurs remercient Aline Maksimov et Carl Sell pour leur excellente assistance technique.




